在过去的研究中,人们已经发现染色质构象对于转录调控非常重要。然而,染色质构象与转录调控之间的关系仍然不明确。近日,东北师范大学生命科学学院张铧坤教授与英国约翰英纳斯中心丁一倞团队合作在Nature Communications上发表了题为A fine-scale Arabidopsis chromatin landscape reveals chromatin conformation-associated transcriptional dynamics的研究论文,利用化学交联辅助的临近捕获 (CAP-C)技术,首次在拟南芥中准确捕获了染色质相互作用,并绘制了分辨率为200bp的染色质互作精细图谱。高分辨率的染色质互作图谱第一次使得研究基因内部染色质构象同转录的关系成为可能。研究结果显示基因内部的局部染色质构象与转录起始、停顿和终止位点的 RNA聚合酶Ⅱ密度显著相关,当局部染色质构象发生变化时,RNA聚合酶Ⅱ活性也会发生相应的变化,表明局部染色质动态变化可能调节 Pol II 活性。此外,该研究还绘制了基因组水平的启动子-启动子相互作用 (PPI) 网络,揭示了PPI 网络负责植物冷胁迫条件下基因表达的共调控机制。
CAP-C是一种高分辨率的染色质互作捕获技术,最早由何川教授团队在果蝇、老鼠胚胎干细胞和人体细胞中创建,此次研究是该方法在植物中的首次应用和延伸。该技术利用补骨脂素 (psoralen) 修饰的聚合物 (polymer) 以不依赖蛋白质互作的方式直接捕获染色质互作信息:其中补骨脂素能直接交联DNA, 捕获近距离染色质相互作用,聚合物上的键击化学 (click-chemistry) 因子同接头 (bridge-linker) 发生键击化学反应,确保捕获的染色质互作的可靠性。与传统的Hi-C方法相比,CAP-C方法减少了对蛋白质-DNA交联的依赖性从而大大提高染色质相互作用分辨率,并且能够更准确地捕获染色质互作信息(图1)。
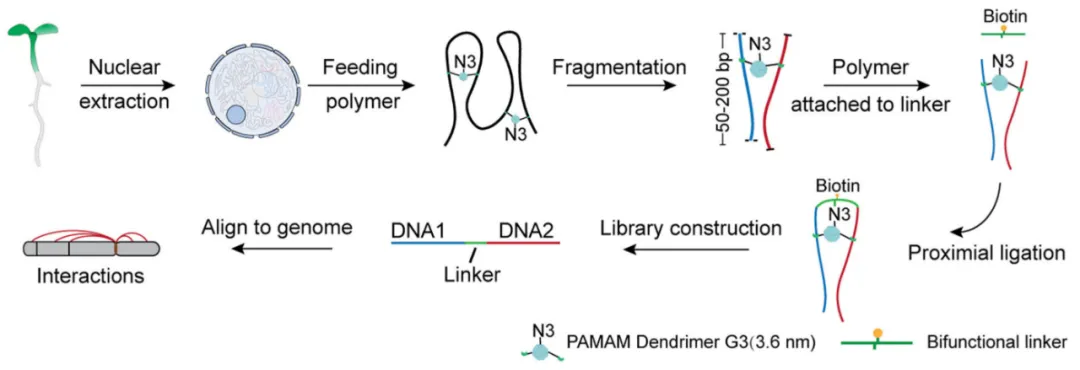
图1:CAP-C文库构建实验流程
通过CAP-C方法,研究人员发现了拟南芥染色质的精细尺度特征,并将其与转录调控相关联。结果显示,染色质相互作用与RNA聚合酶II (Pol II) 活性密切相关;Pol II的停滞现象与局部染色质相互作用之间存在明显的正相关性。
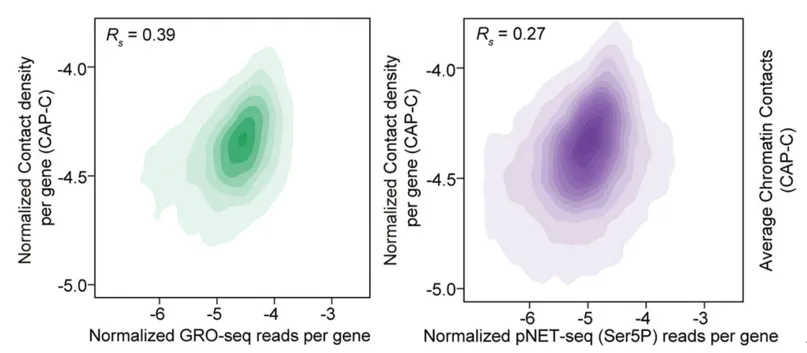
图2: 染色质相互作用与RNA聚合酶II活性及停滞密切相关
进一步研究揭示了植物对冷胁迫响应时染色质构象的变化。当植物在冷胁迫下染色质发生变化时,Pol II的活性也发生了相应的动态变化,表明局部染色质动态可能调节Pol II的活性。
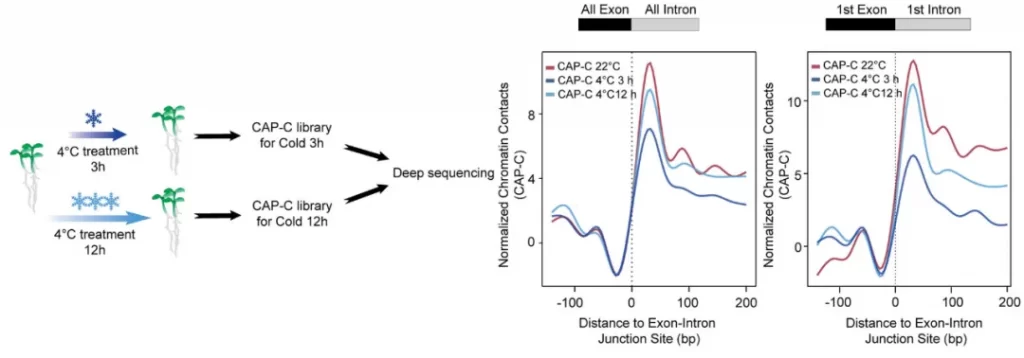
图3:冷胁迫导致染色质相互作用与Pol II活性的动态变化
此外,该研究还鉴定出的全基因组范围的稳定存在的启动子-启动子相互作用网络(PPI)。通过CRIPSR删除相关的启动子区染色质互作导致启动子连接基因的共表达调控消失,证明这些PPI互作网络保证了连接基因的共表达调控,这可能揭示了植物调控转录响应逆境反应的新机制。
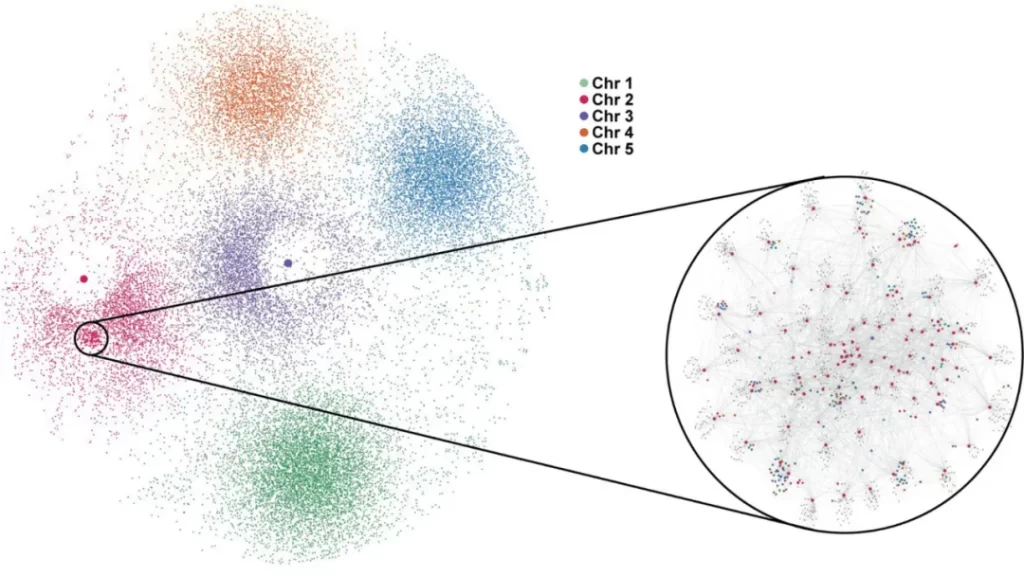
图4:全基因组的启动子-启动子相互作用网络
该论文的第一作者是英国约翰英纳斯中心张月莹博士与东北师范大学董芊里博士、中国科学院遗传与发育生物学研究所王震博士、芝加哥大学刘沁哲博士。通讯作者是东北师范大学生命科学学院张铧坤教授和英国约翰英纳斯中心终身研究员丁一倞。在此,特别要诚挚感谢何川教授在项目实施过程中给予的无私帮助。这个项目的起始就是因为受到了何川教授的启发和指导,没有他的支持与贡献,这个项目就无法顺利展开。感谢曹晓风院士、于昊澎博士、孙文青、Jitender Cheema、游乾承博士和丁玲对于这项工作的贡献。谢忆琳提供了拟南芥激活性增强子pNET-seq数据。Sebastian Marquardt教授为该项目的plaNET-seq数据的分析提供了指导。Caroline Dean院士参与了该项目的讨论。该研究得到了中国国家重点研发计划、中国国家重点研究与发展计划、中国国家自然科学基金、中央高校基本科研业务费、英国生物技术与生物科学研究委员会以及欧洲研究理事会的支持。
论文链接:
https://www.nature.com/articles/s41467-024-47678-7
原文转载自公众号:BioArt植物